TL;DR
- numpy配列にfor文使うと遅い
- ラプラシアンの計算に
scipy.signal.convolve2d使うと早い - でもfor文回すコードに
numba.jitでJITをかけるともっと早い
はじめに
プログラムで何か数値シミュレーションをやることを考える。できるだけ簡単で、かつそれなりに結果が面白いものが良い。わりと簡単なのは熱拡散方程式で、これは定常状態が非常に簡単に求まるのでデバッグがしやすく、かつ非定常状態もフーリエ変換で求まるため、大学生の課題としてはとても良いのだが、何しろ結果が地味なのが泣き所である。
次に面白いのは力学系、特にローレンツ・アトラクタなどが簡単で面白い。しかし、シミュレーションをするので、何かアニメーションを表示させたい。カオスでアニメーションというと二重振り子などがあり、これはこれで面白いのだが、もう一声派手さが欲しい。
というわけで、拡散方程式の次のステップとしては最適なのが反応拡散方程式である。反応拡散方程式とは、その名の通り拡散方程式に反応を表現する力学系がくっついたような模型で、プログラムが非常に簡単なわりに派手な結果が得られ、かつアニメーションもそこそこ面白いので講義の題材としては最適である。
反応拡散系はいろんな種類があるが、私がよく取り上げるのが以下のGray-Scottモデルである。
詳しくは「gray-scott model」とかでググっていただくことにして、簡単に言えば二種類の化学物質がお互いに反応しながら拡散していく様子を表すモデルである。だいたい反応拡散系は活性化因子と抑制因子があり、お互いに反応しながら空間的に複雑なパターンを作っていく。
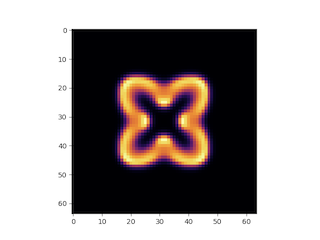
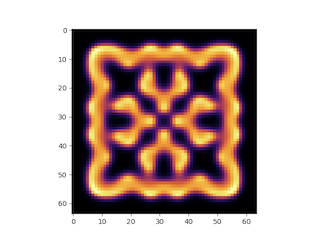
例えば上記の模型なら、こんな時間発展をしていく。
アニメーションもわりと面白いので、興味のある方は試して見られたい。ソースコードは以下に置いてある。
さて、ここまでが長い前振りである。
素直な実装
上記のプログラムを、たとえばPythonで実装することを考えよう。データは二次元のnumpy配列で持つのが自然であろう。力学系の部分はnumpy配列同士の演算で簡単に計算できる。問題はラプラシアンのところである。ラプラシアンの計算には上下左右の四点の平均と、自分との差を計算する必要があるのだが、すなおに実装するとnumpy配列に対してインデックスアクセスしながらfor文を回す必要がある。
例えばこんな感じの実装になろう。
def laplacian(ix, iy, s):
ts = 0.0
ts += s[ix-1, iy]
ts += s[ix+1, iy]
ts += s[ix, iy-1]
ts += s[ix, iy+1]
ts -= 4.0*s[ix, iy]
return ts
def calc(u, v, u2, v2):
(L, _) = u.shape
dt = 0.2
F = 0.04
k = 0.06075
lu = np.zeros((L, L))
lv = np.zeros((L, L))
for ix in range(1, L-1):
for iy in range(1, L-1):
lu[ix, iy] = 0.1 * laplacian(ix, iy, u)
lv[ix, iy] = 0.05 * laplacian(ix, iy, v)
cu = -v*v*u + F*(1.0 - u)
cv = v*v*u - (F+k)*v
u2[:] = u + (lu+cu) * dt
v2[:] = v + (lv+cv) * dt
laplacianは、指定された位置の差分化されたラプラシアンを返す関数、calcは現在のステップの状態u,vから次のステップの状態u2,v2を計算する関数である。力学系部分は素直に実装しているが、ラプラシアンを計算するところはforによる二重ループで実装してある。
初期化を含めた計算は
def main():
L = 64
u = np.zeros((L, L))
u2 = np.zeros((L, L))
v = np.zeros((L, L))
v2 = np.zeros((L, L))
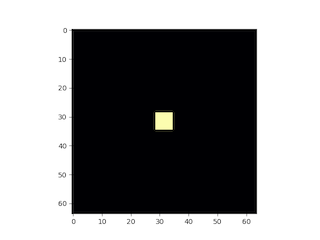
h = L//2
u[h-6:h+6, h-6:h+6] = 0.9
v[h-3:h+3, h-3:h+3] = 0.7
for i in range(10000):
if i % 2 == 0:
calc(u, v, u2, v2)
else:
calc(u2, v2, u, v)
return v
でできる。uとvに正方形の形に初期値を与え、偶数ステップと奇数ステップで入力と出力を入れ替えながら計算を進めていけば良い。64*64を1万ステップ計算している。これをそのまま実行すると、手元のマシンで90秒以上かかる。
$ time python gs.py
python gs.py 92.37s user 0.27s system 99% cpu 1:33.15 total
もともと一週間でなれる!スパコンプログラマという記事の中で反応拡散方程式を取り上げたのだが、その時はC++で書いた。それをPythonに移植したのだが、いくらなんでも遅すぎる。というわけで高速化を考えよう。
scipy.signal.convolve2dを使ってみる
とりあえずnumpyにfor文は遅いというのはわりとよく聞く話である。というわけで別の方法を考える。いくつか候補があるが、例えばscipy.signal.convolve2dを使ってみよう。これはフィルターを自分で定義してコンボリューションを取ることができる。これを使うと計算部分はこうかける。
def calc(u, v, u2, v2):
dt = 0.2
F = 0.04
k = 0.06075
laplacian = np.array([[0, 1, 0], [1, -4, 1], [0, 1, 0]])
lu = 0.1*convolve2d(u, laplacian, mode="same")
lv = 0.05*convolve2d(v, laplacian, mode="same")
cu = -v*v*u + F*(1.0 - u)
cv = v*v*u - (F+k)*v
u2[:] = u + (lu+cu) * dt
v2[:] = v + (lv+cv) * dt
ラプラシアンフィルタを自分で定義し、それを convolve2dに食わせることでラプラシアンを表現している。mode="same"とは、入力と同じ形状で返せ、という意味である。微妙に先程のコードとやってることが違うのだが、多目に見てほしい。
これを実行するとわりと早くなる。
$ time python gs_convolve.py
python gs_convolve.py 3.75s user 0.23s system 93% cpu 4.269 total
4秒弱、先程の素直な実装に比べて25倍近くなった。めでたい。
numba.jitを試す
numpyをfor文回すと遅いなぁ、と思ってたらNumPyでfor文を使うと遅いと思ったがそんな事はなかったぜという記事を見つけた。numba.jitを使うとfor文が早くなる、というものである。早速試してみよう。といってもnumbaをimportして、必要なところに@jitをつけるだけである。
import matplotlib.pyplot as plt
import numpy as np
from numba import jit # 最初にnumba.jitをimportする
@jit # 重い関数の直前に@jitをつける
def laplacian(ix, iy, s):
ts = 0.0
ts += s[ix-1, iy]
ts += s[ix+1, iy]
ts += s[ix, iy-1]
ts += s[ix, iy+1]
ts -= 4.0*s[ix, iy]
return ts
@jit
def calc(u, v, u2, v2):
(L, _) = u.shape
dt = 0.2
F = 0.04
k = 0.06075
lu = np.zeros((L, L))
lv = np.zeros((L, L))
for ix in range(1, L-1):
for iy in range(1, L-1):
lu[ix, iy] = 0.1 * laplacian(ix, iy, u)
lv[ix, iy] = 0.05 * laplacian(ix, iy, v)
cu = -v*v*u + F*(1.0 - u)
cv = v*v*u - (F+k)*v
u2[:] = u + (lu+cu) * dt
v2[:] = v + (lv+cv) * dt
# 以下同文
さて、実行してみよう。
$ time python gs_jit.py
python gs_jit.py 1.75s user 0.25s system 88% cpu 2.265 total
おおー、numba.jitするだけで50倍以上早くなった。これくらい早くなってくれれば現実的な時間でいろいろシミュレーションできて楽しい。
ちなみにscipy.signal.convolve2d版にJITをかけてみたら逆に遅くなった。
まとめ
numpy配列にfor文を回すとそのままでは遅いけど、JITかけると早くなるので、あまり気にしなくてよさそうですね。
コメント